【玉川大学農学部生産農学科 共同研究成果】ヒトの7倍の巨大ゲノムを解読― イベリアトゲイモリが示す発生・再生・進化・行動の謎―
農学部生産農学科の原本悦和教授が参画した「イベリアトゲイモリのゲノム解読」に関する共同研究の成果が、米国の学術雑誌 iScience のオンライン先行版に掲載されました。

有尾両生類であるイモリは、古くから発生や再生の研究において重要な役割を果たしてきました。しかしイモリのゲノムは、反復配列によりヒトの数倍から十数倍と巨大であるため、長らく決定が困難でした。今回、主に国内の研究者からなる「イベリアトゲイモリ研究コンソーシアム」を中心として、日本で樹立された近交系統イベリアトゲイモリ#1を対象に最新の高精度ロングリードシークエンス技術を用いてゲノム解読に成功しました。そのゲノムは約200億塩基対に達し、ヒトの約7倍もの大きさです。解析の結果、ゲノム巨大化に関わる反復配列、器官再生における遺伝子発現制御、両生類の進化やイモリ特有の生殖行動に関わる遺伝子の特徴などが明らかになりました。本研究の成果は、イモリが持つ様々でユニークな生命現象の謎に迫るための重要な情報として、生物学の分野において幅広い展開が期待されます。本研究成果は、2025年9月9日に米国学術雑誌iScienceのオンライン先行版に掲載されました。
本研究成果のポイント
- 反復配列の多さからこれまで解読が困難であった200億塩基というヒトの7倍の大きさを持つイベリアトゲイモリ近交系統のゲノム配列の解読に成功しました。トランスポゾンなどの反復配列により巨大化したゲノムの構成を明らかにしました。
- 他の脊椎動物のゲノム配列と比較を行った結果、有尾両生類のユニークな発生、再生、進化、行動の謎を紐解く遺伝子の特徴を次々と発見しました。
- 脊椎動物の体作りに重要な遺伝子のいくつかが失われていた
- イモリの繁殖行動に適したフェロモン関連遺伝子の進化
- 遺伝子発現を調節するエンハンサー配列が遠く離されても機能している
- 四肢再生能力に関わる可能性のあるゲノム配列の発見
研究の背景
有尾両生類であるイモリは、身近でありながらユニークな特徴を備えた、生物学研究にとって重要な動物の一つです。そのため、古くから発生の仕組みや繁殖行動の謎などを解き明かす研究に貢献してきました。特に、失われたり傷ついたりした組織や器官を元通りに修復する非常に高い器官再生能力を持っており、再生医学の研究対象にもなっています。モデル動物として近年注目されているイベリアトゲイモリは、生物学研究に欠かせない近交系統が日本で樹立維持されています。また国内の一部のメンバーも参加した国際共同研究プロジェクトでは、今年の2月に野生型イベリアトゲイモリの染色体レベルでのゲノム解読を報告しました#2。このイモリを用いた実験を深く広く発展させるためには近交系統のゲノム決定が必要でした。しかしながら、反復配列#3に富んだヒトの約7倍といった非常に大きなゲノム(20G、200億塩基対)#4であるため、技術的な壁が高く、その解読は困難とされていました。
研究の成果
今回、イベリアトゲイモリを用いて研究を行う国内グループ(イベリアトゲイモリ研究コンソーシアム)を中心として、近交系統イベリアトゲイモリのゲノム解読に成功しました。これは、高精度なロングリードシークエンス技術#5の急速な発展により、当初の技術的な問題点を克服できたためです。同じ有尾両生類であるアホロートル(ウーパールーパー)を含む他の脊椎動物のゲノム配列と比較解析したところ、様々なイモリの生物学的特徴が判明しました。その中でも特に興味深い代表的発見を以下に挙げます。
1)巨大ゲノムの大部分を占める反復配列
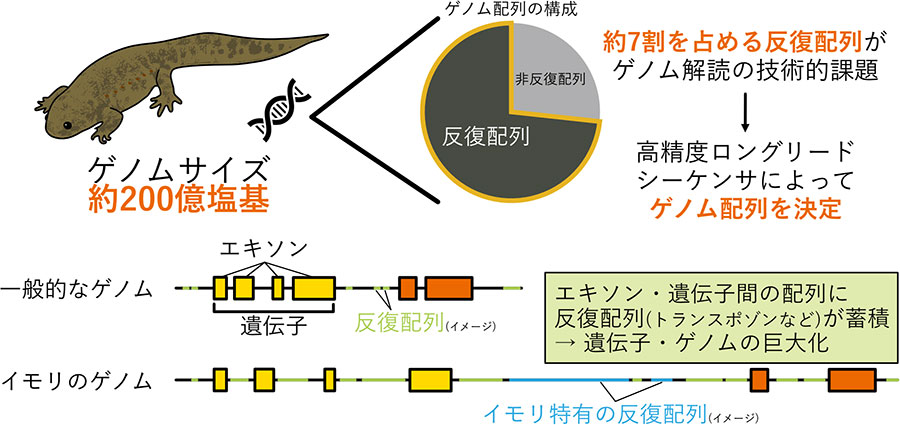
イベリアトゲイモリのゲノム巨大化に関与する反復配列の種類と割合が明らかになりました。イモリのゲノムの約7割が反復配列で占められており、そのほとんどが「トランスポゾン」というゲノム中を動き回る配列でした。反復配列の種類と割合は、巨大ゲノムを持つアホロートルやハイギョとは異なり、ゲノムの巨大化の過程が生物によって異なることがわかりました。また反復配列により、遺伝子のエキソン間、遺伝子間の距離#6、そして遺伝子の転写制御を行うゲノム配列(エンハンサー)と遺伝子間の距離も非常に大きく離れることが分かりました(詳しくは4)のShh遺伝子を参照)。ボディプランに関わる遺伝子にもこのような特徴がありながら、基本的な体の形作りには影響を及ぼしていません。今回新しく特有の反復配列(サテライト配列)を発見しました。この反復配列はイベリアトゲイモリ以外にもアカハライモリ、ブチイモリで確認されており、イモリ属特有と考えられます。
2)有尾両生類の形態形成遺伝子
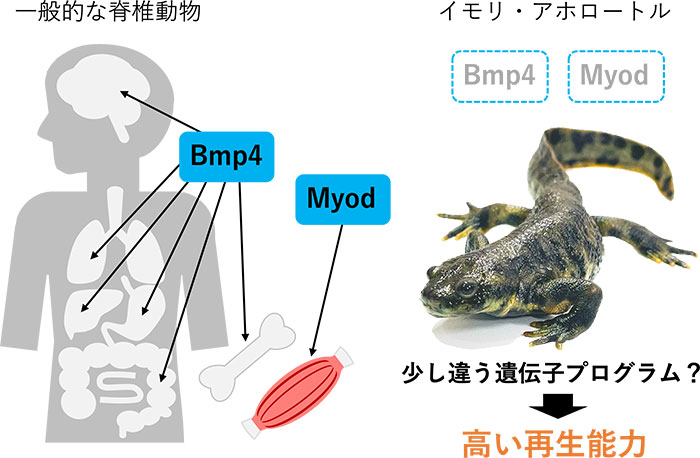
脊椎動物の体の形作りに欠かせない形態形成遺伝子#7のいくつかが、有尾両生類において失われている可能性が高いこともわかりました。Bmp4(骨形成因子4)は脳、眼、内臓、歯や骨など動物の幅広い組織や器官の形成に重要な遺伝子ですが、イモリやアホロートルでは失われている可能性がわかりました。また、MyoD遺伝子は筋肉の形成に重要であり、ヒトを含む他の脊椎動物には必ず存在しています。イモリやアホロートルにも筋肉がありますが、Bmp4と同様にMyoD遺伝子も失われている可能性が高いことが明らかになりました。このことから、進化の過程で他の脊椎動物とは少し違った形態形成遺伝子プログラムを持つようになり、それが高い再生能力にも関連しているのかもしれません。
3)フェロモン遺伝子の変化と繁殖行動の多様性
アカハライモリから発見されたメス誘引フェロモンであるソデフリン#8は、前駆体が切断されて10アミノ酸ほどの短いタンパク質(ペプチド)として機能します。今回の研究でイベリアトゲイモリにおいては前駆体が切断されず、そのままでフェロモンとして機能している可能性が示唆されました。このことはアカハライモリのオスは尻尾を振ってフェロモンを遠くに拡散させてメスを呼び込むのに対し、イベリアトゲイモリのオスはメスを羽交締めするという、繁殖行動の違いに深く関連しているようです。

4)巨大ゲノムにおける遺伝子の転写制御
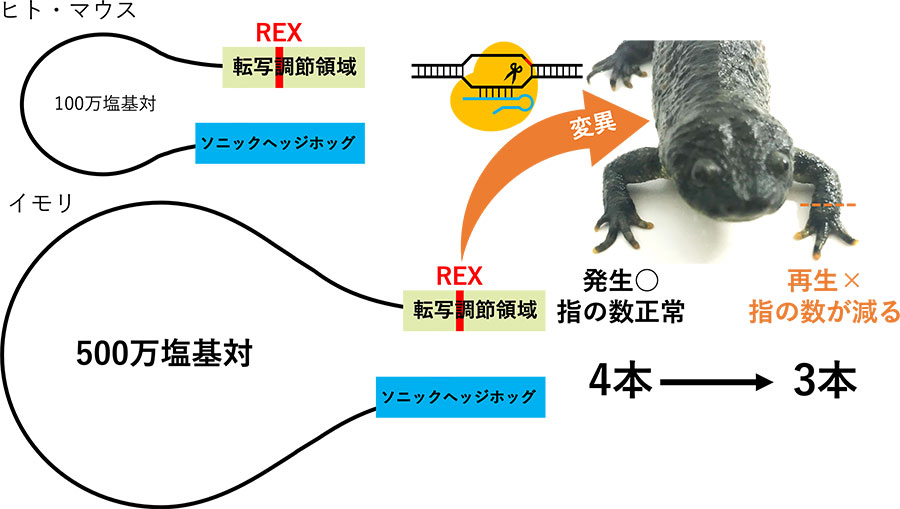
四肢(手足)形成に重要なソニックヘッジホッグ遺伝子(Shh)の転写調節領域(エンハンサー)#9は他の脊椎動物では100万塩基対も遠く離れて機能していることが知られています。有尾両生類ではゲノムの巨大化によってShh遺伝子とエンハンサー間が約500万塩基対も離れているにもかかわらず、正常に機能して四肢が作られることが証明されました。また、この超長距離間で遺伝子発現を正確にオンにするための特殊な配列が存在します。イベリアトゲイモリにおいてゲノム編集#10を用いてこの配列の一つに変異を入れると、四肢発生は正常であるが四肢再生には異常が現れることが判明しました。これは、この配列が再生にも関与している可能性があることを示唆しています。
今後の展開
今回のゲノム解読によって、イモリのゲノム巨大化、発生、再生など様々な謎を解き明かしてきました。巨大ゲノムにおける遺伝子の転写や翻訳、さらには両生類の進化や行動の多様性を知るうえでの重要な情報となります。特にイベリアトゲイモリを用いた再生能力の研究がより広く深く展開されることで、再生医療分野への大きな貢献が期待されます。イベリアトゲイモリはゲノム編集効率が非常に高く、日本で樹立された近交系統イモリリソースを併せることで、イモリを用いた器官再生研究がより加速することが期待されます。
語句説明
- #1 イベリアトゲイモリ
- イベリア半島原産のイモリの一種で学名はPleurodeles waltl。飼育が容易で、一年以内に性成熟(卵や精子を作ることができる成体になること)し、ホルモン注射により一年中受精卵を得ることが可能な動物です。広島大学両生類研究センターのナショナルバイオリソースプロジェクトにて維持管理されています。今回のゲノム解読に用いたイベリアトゲイモリは、二つ番の親から始まって8世代(10年以上)もかけ合わせてできた近交系統です。ゲノム配列の大部分が共通するために個体差が少なく、生物学研究において大変有益なモデル動物です。
- #2 野生型イベリアトゲイモリゲノム解読
- スウェーデンのKarolinska研究所やドイツのMaxPlank-CBG研究所を中心とした国際共同研究グループに基礎生物学研究所の鈴木賢一・広島大学の林利憲・国立遺伝学研究所の豊田敦らも参加し、野生型イベリアトゲイモリのゲノムプロジェクトを行った成果を2025年2月に発表しました(Brown et al., Cell Genomics, 2025; 10.1016/j.xgen.2025.100761)。野生型と今回用いた近交系統イモリでは生息地の由来や進化的に分岐した年数が異なる可能性があり、また高度に近交化した動物のゲノム配列を決定することは比較解析や提供しているリソースの活用上必要であったため、本研究で新規に決定しました。
- #3 反復配列
- ゲノムに存在する反復配列の一種にトランスポゾンがあります。トランスポゾンは自身の配列をゲノム上の別の場所に移動やコピーすることで増えていきます。この反復配列が遺伝子の中(イントロンの中)や遺伝子と遺伝子の間で大量に蓄積された結果、イモリやアホロートル、ハイギョのゲノムは巨大化したと考えられています。
- #4 ゲノムサイズ
- ゲノムサイズとは、生物が持つ 1セットの染色体に含まれるDNAの総量のことで、塩基対数(bp)で表します。ヒトのゲノムサイズは約30億(3G; ギガ)塩基対です。マウスは約2.7G、ニワトリは約1Gとヒトと比べて小さい一方、アホロートル(メキシコサンショウウオ)は32G、ハイギョでは40G(400億)と脊椎動物間でも多様なサイズが存在します。
- #5 高精度ロングリードシークエンス技術
- PacBio社が開発したHiFiリード技術は、ゲノムDNA配列断片を長く正確に配列決定する技術の一つです。この技術によって、反復配列を含む長いゲノムDNA断片も正確に配列を決定できるようになりました。その結果、例えるとピースの大きなパズルを組むように、コンピューター上で大量のシークエンスした断片の配列を繋げる(アセンブルという)ことも容易になりました。イモリのようにゲノムサイズが大きく反復配列を大量に含むゲノム配列を比較的短期間で決定することができたのは、この技術を用いたためです。
- #6 遺伝子(エクソンとイントロン)と転写
- タンパク質に翻訳される配列(エクソン)と、そうではない配列(イントロン)があります。ゲノム上の遺伝子は、このエクソンとイントロンが交互に並んでつながって一つの単位(遺伝子)となります。この遺伝子の配列からmRNAが合成されることを転写といい、このときエクソンとイントロンの両方が一本のmRNAとして転写されます。その後、不要なイントロンは取り除かれ、エクソンだけがつなぎ合わされて、タンパク質として翻訳されます。動物のゲノム上にはこの遺伝子が約2〜3万種類存在しています。翻訳されないイントロンの中や遺伝子と遺伝子の間には反復配列などのDNA配列が大量に蓄積され、ゲノムの巨大化に繋がっていると考えられています。
- #7 形態形成遺伝子
- 形態形成とは、生物個体の発生過程で体の組織や器官(手足や肺や脳など)が形成される現象のことです。形態形成において特定の機能や役割を果たす形態形成遺伝子の異常は、組織や器官の形成不全、機能不全、疾患、腫瘍の原因となります。形態形成遺伝子の有名な例としてはHox、BMP、MyoD、Shh遺伝子などがあります。
- #8 ソデフリン
- 日本の菊山榮博士らがアカハライモリから発見した、オスがメスを惹きつけるフェロモンです。ソデフリンは、アカハライモリでは短いペプチド(10アミノ酸)としてオスの総排泄腔から放出され、尻尾を振って水中で拡散させることによりメスを呼び込みます。その行動が萬葉和歌にうたわれた袖を振る姿に似ていることからソデフリンと名付けられました。
- #9 Shh(ソニックヘッジホッグ)遺伝子と遺伝子の転写
- 動物の様々な組織や器官の形成に必須の有名な形態形成遺伝子の一つです。また、転写調節領(エンハンサー)とは、遺伝子発現(転写、mRNA合成)を適切にオンオフするために必要なゲノム配列のことです。四肢脊椎動物において、Shh遺伝子の転写調節領域であるMFCS1/ZRSエンハンサーに変異が生じると、ヒトを含む四肢脊椎動物の指が増えたり減ったりする現象が起こります(多肢症や四肢の短縮を含む疾患)。このMFCS1/ZRSには長距離でエンハンサーを作用させる機能を持つ特殊な配列(REX)が含まれていることが報告されています(Bower et al., Nature, 2025)。イモリのMFCS1/ZRSでもこの配列REXが保存されており、本研究ではゲノム編集によって同配列の一つに変異を入れたイモリを解析しました。
- #10ゲノム編集
- CRISPR/Cas9に代表される人工DNA切断酵素によってゲノムDNAにDNA二本鎖切断を誘導した際に、その修復過程において標的遺伝子への欠失や挿入変異を挿入し、遺伝子の機能を破壊したり改変したりする技術のことです。
論文情報
- 掲載雑誌名:
iScience(Cell Press)
- 掲 載 日:
2025年9月9日
- DOI :
- タイトル :
“The inbred newt genome unveils molecular mechanisms of behavior, development, and regeneration in urodele amphibians”
- 著 者:
Yuki Kimura1†,Miyuki Suzuki2†,Akinori Okumura3†,Masatoshi Matsunami4†,Hiroyo Nishide3†,Rima Mizuno5,Kazuto Bou5,Yoshinobu Uno6,Tomoaki Nakada7,Itaru Hasunuma8,Yoshikazu Haramoto9,Akimasa Fukui10,Takeshi Inoue11,Yuki Sato12,Katsushi Yamaguchi3,Zicong Zhang13,Akane Chihara3,Mai Takehara14,Yuki Shibata15,Masaaki Kitada12,Nerea Moreno16,Ikuo Uchiyama3,Yutaka Suzuki17,Takashi Takeuchi11,Masato Nikaido1,Kiyokazu Agata3,Atsushi Toyoda18,Shuji Shigenobu3,Toshinori Hayashi14*,Ken-ichi T Suzuki3*
- 所 属:
- 1.
東京科学大学(旧・東京工業大学): †木村優希・二階堂雅人
- 2.
カリフォルニア工科大学(元・基礎生物学研究所):†#鈴木美有紀
- 3.
基礎生物学研究所(5.総合研究大学院大学):†奥村晃成・†#西出浩世・水野莉万・保和人・#山口勝司・千原あかね・#内山郁夫・#阿形清和・#重信秀治・*#鈴木賢一
- 4.
琉球大学:†#松波雅俊
- 6.
徳島大学:宇野好宣
- 7.
日本獣医生命科学大学:中田友明
- 8.
東邦大学:蓮沼至
- 9.
玉川大学:#原本悦和
- 10.
中央大学:#福井彰雅
- 11.
鳥取大学:#井上武・#竹内隆
- 12.
関西医科大学:佐藤勇輝・北田容章
- 13.
京都大学:Zicong Zhang
- 14.
広島大学:竹原舞・*#林利憲
- 15.
日本医科大学(元・基礎生物学研究所):柴田侑毅
- 16.
マドリード・コンプルテンセ大学:Nerea Moreno
- 17.
東京大学(先進ゲノム支援):鈴木穣
- 18.
国立遺伝学研究所(先進ゲノム支援):豊田敦
†は筆頭著者五名、*は共同責任著者、#はイベリアトゲイモリ研究コンソーシアム
- 1.
研究グループ
本研究は基礎生物学研究所 超階層生物学センター 新規モデル生物開発室の鈴木賢一と広島大学 両生類研究センターの林利憲がプロジェクト責任者として、基礎生物学研究所 超階層生物学センター トランスオミクス解析室の重信秀治らと国立遺伝学研究所(先進ゲノム支援)の豊田敦らの協力によりゲノムシークエンスとアセンブルを行いました。次に、東京科学大学の木村優希(元 二階堂雅人研究室)、カリフォルニア工科大学HFSPフェローの鈴木美有紀(元 基礎生物学研究所)、琉球大学大学院医学研究科の松波雅俊、そして基礎生物学研究所 超階層生物学センター 新規モデル生物開発室の奥村晃成とデータ統合解析室の西出浩世らを中心として、イベリアトゲイモリ研究コンソーシアムメンバーと他大学機関のイベリアトゲイモリ研究者が加わり詳細な解析を行いました。
研究サポート
本研究は、基礎生物学研究所 新規モデル生物共同利用研究、文部科学省 科学研究費助成事業 学術変革領域研究 学術研究支援基盤形成(先進ゲノム支援 [PAGS])、日本学術振興会 科学研究費助成事業、国立研究開発法人科学技術振興機構・戦略的創造研究推進事業(JST-CREST)、文部科学省 ナショナルバイオリソースプロジェクトをはじめとする関係機関のご支援のもとに行われました。






